Bieżący numer
O czasopiśmie
Rada Naukowa
Kolegium Redakcyjne
Polityka prawno-archiwizacyjna
Kodeks etyki publikacyjnej
Wydawca
Informacja o przetwarzaniu danych osobowych w ramach plików cookies oraz subskrypcji newslettera
Archiwum
Dla autorów
Dla recenzentów
Kontakt
Recenzenci
Recenzenci rocznika 2025
Recenzenci rocznika 2024
Recenzenci rocznika 2023
Recenzenci rocznika 2022
Recenzenci rocznika 2021
Recenzenci rocznika 2020
Recenzenci rocznika 2019
Recenzenci rocznika 2018
Recenzenci rocznika 2017
Recenzenci rocznika 2016
Recenzenci rocznika 2015
Recenzenci rocznika 2014
Recenzenci rocznika 2013
Recenzenci rocznika 2012
Polecamy
Śląski Uniwersytet Medyczny w Katowicach
Sklep Wydawnictw SUM
Biblioteka Główna SUM
Polityka prywatności
Deklaracja dostępności
Recenzenci
Recenzenci rocznika 2025
Recenzenci rocznika 2024
Recenzenci rocznika 2023
Recenzenci rocznika 2022
Recenzenci rocznika 2021
Recenzenci rocznika 2020
Recenzenci rocznika 2019
Recenzenci rocznika 2018
Recenzenci rocznika 2017
Recenzenci rocznika 2016
Recenzenci rocznika 2015
Recenzenci rocznika 2014
Recenzenci rocznika 2013
Recenzenci rocznika 2012
Analiza mikrobiomu jelitowego otyłych małżonków chorujących na cukrzycę – opis przypadku i przegląd literatury
1
Department of Internal Diseases Propaedeutics and Emergency Medicine, Faculty of Public Health in Bytom, Medical University of Silesia, Katowice, Poland
2
University of Applied Sciences in Nysa, Poland
Autor do korespondencji
Magdalena Piłot
Katedra i Zakład Propedeutyki Chorób Wewnętrznych i Medycyny Ratunkowej, Wydział Zdrowia Publicznego w Bytomiu, Śląski Uniwersytet Medyczny w Katowicach, ul. Piekarska 18, 41-902 Bytom
Katedra i Zakład Propedeutyki Chorób Wewnętrznych i Medycyny Ratunkowej, Wydział Zdrowia Publicznego w Bytomiu, Śląski Uniwersytet Medyczny w Katowicach, ul. Piekarska 18, 41-902 Bytom
Ann. Acad. Med. Siles. 2024;78:31-40
SŁOWA KLUCZOWE
DZIEDZINY
STRESZCZENIE
Wstęp:
Zmiany składu mikrobiomu jelitowego odgrywają istotną rolę w patofizjologii wielu schorzeń, w tym cukrzycy typu 2. Celem pracy była ocena profilu mikrobiomu jelitowego małżeństwa z otyłością i cukrzycą typu 2, mieszkającego 35 lat we wspólnym gospodarstwie domowym, pod względem stanu odżywienia, stylu życia i metod leczenia cukrzycy. Jednocześnie podjęto próbę odpowiedzi na pytanie, które czynniki mają największy wpływ na ewentualną dysbiozę jelitową.
Materiał i metody:
Analizie poddano wyniki pomiarów antropometrycznych pacjentów, skład ich ciała, 24-godzinny wywiad żywieniowy, profil glikemii i próbki kału. Do ilościowego i jakościowego badania flory jelitowej w kale zastosowano metodę sekwencjonowania nowej generacji.
Wyniki:
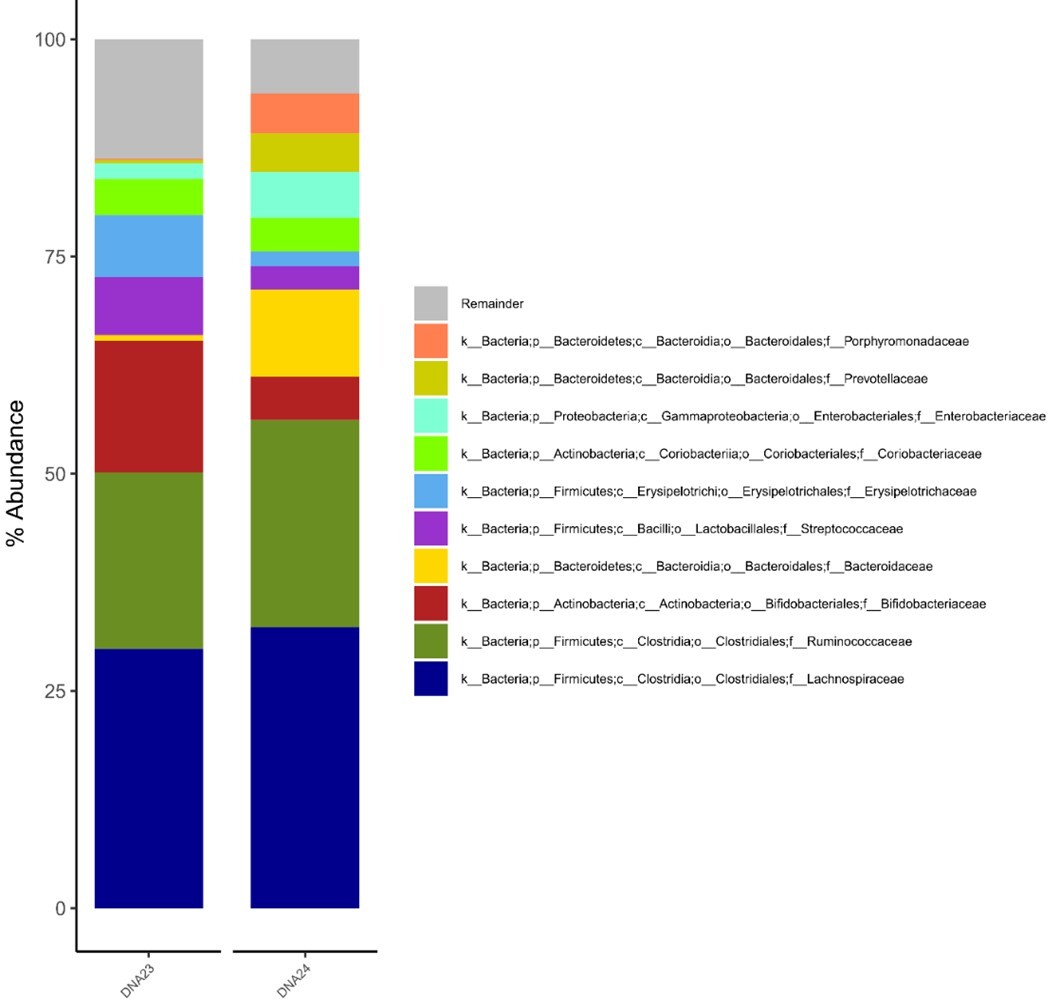
Nie stwierdzono znaczących różnic w badaniu mikrobiomu jelitowego pomiędzy małżonkami. Dominującymi gromadami bakterii były Firmicutes i Actinobacteria, podczas gdy Bacteroidetes i Proteobacteria występowały w proporcjach od 2 do 7%. Gromada Firmicutes była reprezentowana przez dominującą rodzinę Lachnospiraceae (29–31%), Ruminococcaceae (16–19%) i Streptococcaceae (3–11%). Gromada Actinobacteria była proporcjonalnie mniej liczna i reprezentowana głównie przez Bifidobacteriaceae (6–12%).
Wnioski:
Być może wspólne warunki życia mają najbardziej istotny wpływ na skład mikroflory jelitowej małżonków chorych na cukrzycę, mimo różnic dotyczących płci, schorzeń współistniejących, terapii cukrzycy, stosowanej diety i zachowań zdrowotnych.
Zmiany składu mikrobiomu jelitowego odgrywają istotną rolę w patofizjologii wielu schorzeń, w tym cukrzycy typu 2. Celem pracy była ocena profilu mikrobiomu jelitowego małżeństwa z otyłością i cukrzycą typu 2, mieszkającego 35 lat we wspólnym gospodarstwie domowym, pod względem stanu odżywienia, stylu życia i metod leczenia cukrzycy. Jednocześnie podjęto próbę odpowiedzi na pytanie, które czynniki mają największy wpływ na ewentualną dysbiozę jelitową.
Materiał i metody:
Analizie poddano wyniki pomiarów antropometrycznych pacjentów, skład ich ciała, 24-godzinny wywiad żywieniowy, profil glikemii i próbki kału. Do ilościowego i jakościowego badania flory jelitowej w kale zastosowano metodę sekwencjonowania nowej generacji.
Wyniki:
Nie stwierdzono znaczących różnic w badaniu mikrobiomu jelitowego pomiędzy małżonkami. Dominującymi gromadami bakterii były Firmicutes i Actinobacteria, podczas gdy Bacteroidetes i Proteobacteria występowały w proporcjach od 2 do 7%. Gromada Firmicutes była reprezentowana przez dominującą rodzinę Lachnospiraceae (29–31%), Ruminococcaceae (16–19%) i Streptococcaceae (3–11%). Gromada Actinobacteria była proporcjonalnie mniej liczna i reprezentowana głównie przez Bifidobacteriaceae (6–12%).
Wnioski:
Być może wspólne warunki życia mają najbardziej istotny wpływ na skład mikroflory jelitowej małżonków chorych na cukrzycę, mimo różnic dotyczących płci, schorzeń współistniejących, terapii cukrzycy, stosowanej diety i zachowań zdrowotnych.
REFERENCJE (48)
1.
Mithieux G. Gut microbiota and host metabolism: what relationship. Neuroendocrinology 2018; 106(4): 352–356, doi: 10.1159/000484526.
2.
Zhu B., Wang X., Li L. Human gut microbiome: the second genome of body. Protein Cell 2010; 1(8): 718–725, doi: 10.1007/s13238-010-0093-z.
3.
Li Q., Zhou J.M. The microbiota-gut-brain axis and its potential therapeutic role in autism spectrum disorder. Neuroscience 2016; 324: 131–139, doi: 10.1016/j.neuroscience.2016.03.013.
4.
Hattori M., Taylor T.D. The human intestinal microbiome: a new frontier of human biology. DNA Res. 2009; 16(1): 1–12, doi: 10.1093/dnares/dsn033.
5.
Giuffrè M., Campigotto M., Campisciano G., Comar M., Crocè L.S. A story of liver and gut microbes: how does the intestinal flora affect liver disease? A review of the literature. Am. J. Physiol. Gastrointest. Liver Physiol. 2020; 318(5): G889–G906, doi: 10.1152/ajpgi.00161.2019.
6.
Le Chatelier E., Nielsen T., Qin J., Prifti E., Hildebrand F., Falony G. et al. Richness of human gut microbiome correlates with metabolic markers. Nature 2013; 500(7464): 541–546, doi: 10.1038/nature12506.
7.
Giuffrè M., Moretti R., Campisciano G., da Silveira A.B.M., Monda V.M., Comar M., Di Bella S. et al. You talking to me? Says the enteric nerv-ous system (ENS) to the microbe. How intestinal microbes interact with the ENS. J. Clin. Med. 2020; 9(11): 3705, doi: 10.3390/jcm9113705.
8.
Clements S.J., Carding S.R. Diet, the intestinal microbiota, and immune health in aging. Crit. Rev. Food Sci. Nutr. 2018; 58(4): 651–661, doi: 10.1080/10408398.2016.1211086.
9.
Halmos T., Suba I. Physiological patterns of intestinal microbiota. The role of dysbacteriosis in obesity, insulin resistance, diabetes and metabolic syndrome. Orv. Hetil. 2016; 157(1): 13–22, doi: 10.1556/650.2015.30296.
10.
Delzenne N.M., Cani P.D., Everard A., Neyrinck A.M., Bindels L.B. Gut microorganisms as promising targets for the management of type 2 diabetes. Diabetologia 2015; 58(10): 2206–2217, doi: 10.1007/s00125-015-3712-7.
11.
Aydin Ö., Nieuwdorp M., Gerdes V. The gut microbiome as a target for the treatment of type 2 diabetes. Curr. Diab. Rep. 2018; 18(8): 55, doi: 10.1007/s11892-018-1020-6.
12.
Malczyk E., Dzięgielewska-Gęsiak S., Fatyga E., Ziółko E., Kokot T., Muc-Wierzgoń M. Body composition in healthy older persons: role of the ratio of extracellular/total body water. J. Biol. Regul. Homeost. Agents 2016; 30(3): 767–772.
13.
Szponar L., Wolnicka K., Rychlik E. Album of photographs of food products and dishes. National Food and Nutrition Institute. Warszawa 2000.
14.
Normy żywienia dla populacji Polski. M. Jarosz [ed.]. Instytut Żywności i Żywienia. Warszawa 2017.
15.
Jeżewska‐Zychowicz M., Gawęcki J., Wądołowska L., Czarnocińska J., Galiński G., Kołłajtis‐Dołowy A. et al. Kwestionariusz do badania poglądów i zwyczajów żywieniowych dla osób w wieku od 16 do 65 lat, wersja 1.2 – kwestionariusz do samodzielnego wypełnienia przez Respondenta. In: Kwestionariusz do badania poglądów i zwyczajów żywieniowych oraz procedura opracowania danych. J. Gawęcki [ed.]. Komitet Nauki o Żywieniu Człowieka Polskiej Akademii Nauk. Warszawa 2014, pp 21–33. Available at http://www.knozc.pan.pl/.
16.
Klindworth A., Preusse E., Schweer T., Peplies J., Quast C., Horn M. et al. Evaluation of general 16S ribosomal RNA gene PCR primers for classical and next-generation sequencing-based diversity studies. Nucleic Acid. Res. 2013; 41(1): e1, doi: 10.1093/nar/gks808.
17.
Parks D.H., Tyson G.W., Hugenholtz P., Beiko R.G. STAMP: Statistical analysis of taxonomic and functional profiles. Bioinformatics 2014; 30(21): 3123–3124, doi: 10.1093/bioinformatics/btu494.
18.
Stackebrandt E. The Family Lachnospiraceae. In: The Prokaryotes: Firmicutes and Tenericutes. E. Rosenberg, E.F. DeLong, S. Lory, E. Stackebrandt, F. Thompson [ed.]. Springer. Berlin, Heidelberg 2014, pp 197–201, doi: 10.1007/978-3-642-30120-9_363.
19.
Dzięgielewska-Gęsiak S., Fatyga E., Piłot M., Wierzgoń A., Muc-Wierzgoń M. Are there differences in gut microbiome in patients with type 2 diabetes treated by metformin or metformin and insulin? Diabetes Metab. Syndr. Obes. 2022; 15: 3589–3599, doi: 10.2147/DMSO.S377856.
20.
Rinninella E., Raoul R., Cintoni M., Franceschi F., Miggiano G.A.D., Gasbarrini A. et al. What is the healthy gut microbiota composition? A changing ecosystem across age, environment, diet, and diseases. Microorganisms 2019; 7(1): 14, doi: 10.3390/microorganisms7010014.
21.
Ley R.E., Turnbaugh P.J., Klein S., Gordon J.I. Microbial ecology: human gut microbes associated with obesity. Nature 2006; 444(7122): 1022–1023, doi: 10.1038/4441022a.
22.
Arumugam M., Raes J., Pelletier E., Le Paslier D., Yamada T., Mende D.R. et al. Enterotypes of the human gut microbiome. Nature 2011; 473(7346): 174–180, doi: 10.1038/nature09944.
23.
Vacca M., Celano G., Calabrese F.M., Portincasa P., Gobbetti M., De Angelis M. The controversial role of human gut Lachnospiraceae. Microorganisms 2020; 8(4): 573, doi: 10.3390/microorganisms8040573.
24.
Zeng H., Ishaq S.L., Zhao F.Q., Wright A.D.G. Colonic inflammation accompanies an increase of β-catenin signaling and Lachnospiraceae/Streptococcaceae bacteria in the hind gut of high-fat diet-fed mice. J. Nutr. Biochem. 2016; 35: 30–36, doi: 10.1016/j.jnutbio.2016.05.015.
25.
Fei N., Zhao L. An opportunistic pathogen isolated from the gut of an obese human causes obesity in germfree mice. ISME J. 2013; 7(4): 880–884, doi: 10.1038/ismej.2012.153.
26.
Takagi T., Naito Y., Kashiwagi S., Uchiyama K., Mizushima K., Kamada K. et al. Changes in the gut microbiota are associated with hypertension, hyperlipidemia, and type 2 diabetes mellitus in Japanese subjects. Nutrients 2020; 12(10): 2996, doi: 10.3390/nu12102996.
27.
Salonen A., Lahti L., Salojärvi J., Holtrop G., Korpela K., Duncan S.H. et al. Impact of diet and individual variation on intestinal microbiota composition and fermentation products in obese men. ISME J. 2014; 8(11): 2218–2230, doi: 10.1038/ismej.2014.63.
28.
Hashimoto Y., Hamaguchi M., Kaji A., Sakai R., Osaka T., Inoue R. et al. Intake of sucrose affects gut dysbiosis in patients with type 2 diabetes. J. Diabetes Investig. 2020; 11(6): 1623–1634, doi: 10.1111/jdi.13293.
29.
Qin J., Li Y., Cai Z., Li S., Zhu J., Zhang F. et al. A metagenome-wide association study of gut microbiota in type 2 diabetes. Nature 2012; 490(7418): 55–60, doi: 10.1038/nature11450.
30.
Adachi K., Sugiyama T., Yamaguchi Y., Tamura Y., Izawa S., Hijikata Y. et al. Gut microbiota disorders cause type 2 diabetes mellitus and homeostatic disturbances in gut-related metabolism in Japanese subjects. J. Clin. Biochem. Nutr 2019; 64(3): 231–238, doi: 10.3164/jcbn.18-101.
31.
Santos-Marcos J.A., Rangel-Zuñiga O.A., Jimenez-Lucena R., Quintana-Navarro G.M., Garcia-Carpintero S., Malagon M.M. et al. Influence of gender and menopausal status on gut microbiota. Maturitas 2018; 116: 43–53, doi: 10.1016/j.maturitas.2018.07.008.
32.
Larsen N., Vogensen F.K., van den Berg F.W.J., Nielsen D.S., Andreasen A.S., Pedersen B.K. et al. Gut microbiota in human adults with type 2 diabetes differs from non-diabetic adults. PLoS One 2010; 5(2): e9085, doi: 10.1371/journal.pone.0009085.
33.
de la Cuesta-Zuluaga J., Mueller N.T., Corrales-Agudelo V., Velásquez-Mejía E.P., Carmona J.A., Abad J.M. et al. Metformin is associated with higher relative abundance of mucin-degrading Akkermansia muciniphila and several short-chain fatty acid-producing microbiota in the gut. Diabetes Care 2017; 40(1): 54–62, doi: 10.2337/dc16-1324.
34.
Elbere I., Kalnina I., Silamikelis I., Konrade I., Zaharenko L., Sekace K. et al. Association of metformin administration with gut microbiome dysbiosis in healthy volunteers. PLoS One 2018; 13(9): e0204317, doi: 10.1371/journal.pone.0204317.
35.
Wang H., Tang W., Zhang P., Zhang Z., Jielei H., Zhu D. et al. Modulation of gut microbiota contributes to effects of intensive insulin therapy on intestinal morphological alteration in high-fat-diet-treated mice. Acta Diabetol. 2020; 57(4): 455–467, doi: 10.1007/s00592-019-01436-0.
36.
Savin Z., Kivity S., Yonath H., Yehuda S. Smoking and the intestinal microbiome. Arch. Microbiol. 2018; 200(5): 677–684, doi: 10.1007/s00203-018-1506-2.
37.
Sublette M.G., Cross T.L., Korcarz C.E., Hansen K.M., Murga-Garrido S.M., Hazen S.L. et al. Effects of smoking and smoking cessation on the intestinal microbiota. J. Clin. Med. 2020; 9(9): 2963, doi: 10.3390/jcm9092963.
38.
Lee S.H., Yun Y., Kim S.J., Lee E.J., Chang Y., Ryu S. et al. Association between cigarette smoking status and composition of gut microbiota: population-based cross-sectional study. J. Clin. Med. 2018; 7(9): 282, doi: 10.3390/jcm7090282.
39.
David L.A., Maurice C.F., Carmody R.N., Gootenberg D.B., Button J.E., Wolfe B.E. et al. Diet rapidly and reproducibly alters the human gut microbiome. Nature 2014; 505(7484): 559–563, doi: 10.1038/nature12820.
40.
Scott K.P., Gratz S.W., Sheridan P.O., Flint H.J., Duncan S.H. The influence of diet on the gut microbiota. Pharmacol. Res. 2013; 69(1): 52–60, doi: 10.1016/j.phrs.2012.10.020.
41.
Magnúsdóttir S., Ravcheev D., de Crécy-Lagard V., Thiele I. Systematic genome assessment of B-vitamin biosynthesis suggests co-operation among gut microbes. Front. Genet. 2015; 6: 148, doi: 10.3389/fgene.2015.00148.
42.
Ojo O., Feng Q.Q., Ojo O.O., Wang X.H. The role of dietary fibre in modulating gut microbiota dysbiosis in patients with type 2 diabetes: a systematic review and meta-analysis of randomised controlled trials. Nutrients 2020; 12(11): 3239, doi: 10.3390/nu12113239.
43.
Makki K., Deehan E.C., Walter J., Bäckhed F. The impact of dietary fiber on gut microbiota in host health and disease. Cell Host Microbe 2018; 23(6): 705–715, doi: 10.1016/j.chom.2018.05.012.
44.
Schwiertz A., Taras D., Schäfer K., Beijer S., Bos N.A., Donus C. et al. Microbiota and SCFA in lean and overweight healthy subjects. Obesity 2010; 18(1): 190–195, doi: 10.1038/oby.2009.167.
45.
Herd P., Schaeffer N.C., DiLoreto K., Jacques K., Stevenson J., Rey F. et al. The influence of social conditions across the life course on the human gut microbiota: a pilot project with the Wisconsin Longitudinal Study. J. Gerontol. B. Psychol. Sci. Soc. Sci. 2017; 73(1): 124–133, doi: 10.1093/geronb/gbx029.
46.
Dic-McFarland K.A., Tang Z.Z., Kemis J.H., Kerby R.L., Chen G., Palloni A. et al. Close social relationships correlate with human gut microbiota composition. Sci. Rep. 2019; 24; 9(1): 703, doi: 10.1038/s41598-018-37298-9.
47.
Bleau C., Karelis A.D., St-Pierre D.H., Lamontagne L. Crosstalk between intestinal microbiota, adipose tissue and skeletal muscle as an early event in systemic low-grade inflammation and the development of obesity and diabetes. Diabetes Metab. Res. Rev. 2015; 31(6): 545–561, https://doi.org/10.1002/dmrr.2....
48.
Wu G.D., Chen J., Hoffmann C., Bittinger K., Chen Y.Y., Keilbaugh S.A. et al. Linking long-term dietary patterns with gut microbial enterotypes. Science 2011; 334(6052): 105–108, doi: 10.1126/science.1208344.
CYTOWANIA (1):
1.
Gut Microbiota and Metabolic Dysregulation in Elderly Diabetic Patients: Is There a Gender-Specific Effect
Magdalena Piłot, Sylwia Dzięgielewska-Gęsiak, Katarzyna Weronika Walkiewicz, Martyna Bednarczyk, Dariusz Waniczek, Małgorzata Muc-Wierzgoń
Journal of Clinical Medicine
Magdalena Piłot, Sylwia Dzięgielewska-Gęsiak, Katarzyna Weronika Walkiewicz, Martyna Bednarczyk, Dariusz Waniczek, Małgorzata Muc-Wierzgoń
Journal of Clinical Medicine
Udostępnij
ARTYKUŁ POWIĄZANY
Śląski Uniwersytet Medyczny w Katowicach, jako Operator Serwisu annales.sum.edu.pl, przetwarza dane osobowe zbierane podczas odwiedzania Serwisu. Realizacja funkcji pozyskiwania informacji o Użytkownikach i ich zachowaniu odbywa się poprzez dobrowolnie wprowadzone w formularzach informacje, zapisywanie w urządzeniach końcowych plików cookies (tzw. ciasteczka), a także poprzez gromadzenie logów serwera www, będącego w posiadaniu Operatora Serwisu. Dane, w tym pliki cookies, wykorzystywane są w celu realizacji usług zgodnie z Polityką prywatności.
Możesz wyrazić zgodę na przetwarzanie danych w tych celach, odmówić zgody lub uzyskać dostęp do bardziej szczegółowych informacji.
Możesz wyrazić zgodę na przetwarzanie danych w tych celach, odmówić zgody lub uzyskać dostęp do bardziej szczegółowych informacji.